Smerovaná evolúcia
Smerovaná evolúcia je označenie pre technológiu kombinujúcu samovoľné evolučné procesy s ich smerovaním intelegentným činiteľom. Hoci šľachtiteľstvo zvierat a rastlín tiež patrí a zarádza sa k nej, tento názov sa používa hlavne pre biochemické procesy. Má široký záber využitia. Umožňuje či už skúmať a pochopiť procesy prebiehajúce v prírode alebo vytvárať nové biozlúčeniny či aj „organizmy“. Zameriava sa najprv mutáciami vyrobiť rozličné varianty organizmu či zlúčeniny, na selekciu variantov s chcenými parametrami a výrobu „populácie“ pre vylepšenie parametrov variantov – ale vo veľmi veľa opakovaniach.[1]
Mechanizmus fungovania
[upraviť | upraviť zdroj]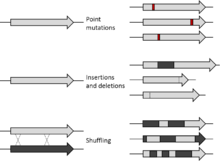
Celý princíp nie je zložitý. Vezmú sa rôzne varianty látky, organizmu, čo máme zámer „vylepšiť“.
- Tieto sa v prvom stupni cyklu (generácie) vystavia mutáciám – zvyčajne náhodným, aby sa „objavili“ varianty s lepšími chcenými parametrami.
- V stupni 2 rôznymi spôsobmi určíme „fitness“ variantov – čím vyššie fitness, tým lepší chcený parameter.
- Varianty s určitým fitness (najvyššími) vezmeme a priebeh sa zopakuje.
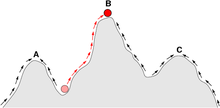
Varianty v x-tej „generácii“ majú lepšie chcené parametre než prvotné. Generácií ale treba aj niekoľko tisíc - záleží na parametroch variantov, „šťastí“, variantoch, vybavení experimentátorov... Veľmi podobne sa môžu meniť varianty aj v prírode – líši sa ale výrazne v tom, že cieľ (parameter) určí inteligentný agent – experimentátor. Prežiť variantom vyberá on – v „smere“ cieľa. Trvanie záleží na automatizácii laboratória, generáciách, mutáciách aj výbere prvej populácie.[2][3]
Využitie a limity
[upraviť | upraviť zdroj]
Býva využívaná na rôzne účely. Či sú to „len“ biochemické zlúčeniny-bielkoviny, a iné alebo organizmy-vírusy, baktérie a iné. Touto metódou môžeme získavať aj látky aj organizmy a časti organizmov s chcenými parametrami pre laboratóriá, medicínu, priemysel, farmáciu a výskum. (Bol vytvorený umelý vírus s veľmi slabým fitness. Po vystavení selekčnému tlaku a niekoľkých cykloch sa jeho fitness zlepšilo rádovo.[5]) Stále ale zostáva kľúčovým prvkom človek. Najnovšie biochemické štúdie tvrdia, že v „mikro“ merítku (na bunkovej alebo genetickej rovine) je na dostatočne pravdepodobný prechod medzi jednotlivými životaschopnými vývojovými štádiami potrebných niekoľko menších krokov. Tento problém je dobre analyzovateľný pri bielkovinách - základných stavebných a funkčných prvkoch organizmov. Každá z nich plní nejakú (zvyčajne kľúčovú) funkciu v organizme. Premena jednej z nich na nejakú novú, ktorá by priniesla organizmu výhodu, vyžaduje niekoľko mutácií. Tieto prechodné formy sú však v absolútnej väčšine nevýhodné alebo priamo likvidačné pre biologický systém. Výhodu pre organizmus prinesie až kompletný počet týchto postupných zmien. Prírodný výber ich teda nemôže podporiť, kým neprinesú nejakú výhodu.[6] [7] Aj v prípade, že tieto prechodné formy sú len neutrálne (neznevýhodňujú organizmus), podrobná analýza ukazuje, že je potrebné extrémne veľké množstvo času, kým sa tieto kroky uskutočnia a zafixujú v „nepestovanej“ populácii.[8] Očakávalo sa, že ak sa použije namiesto postupnosti jednobodových mutácií radšej väčšie množstvo mutácií naraz, či už vedľa seba alebo na rôznych miestach v bielkovine, podarí sa vyhnúť problému nevýhodných prechodných foriem. V súčasnosti však panuje pomerná zhoda v názore, že takéto „mutačné dávky“ vedú k zničeniu funkcie bielkoviny.[9] [10] V poslednej dobe boli preskúmané ďalšie nádejné hypotézy generovania nových bielkovín: náhodná rekombinácia častí proteínu (respektíve ich posun) a vznik nového funkčného génu náhodným vložením časti DNA do intrónu. Výsledky štúdií predkladajú záver, že obidva scenáre sú slepou uličkou a len sa zvýraznila nedostatočnosť čisto náhodných procesov vytvoriť nový funkčný proteín v rámci dosiaľ uvažovaných evolučných rámcov.[11] [12] [13]
Biológia v tomto prípade významne využíva i výsledky informatiky. Pravdepodobne jediným kandidátom, ktorý by bol porovnateľný s mechanizmami akceptovanými biológmi sú tzv. evolučné algoritmy. Používajú simuláciu procesov využívaných evolučnými teóriami (mutácia, kríženie, selekcia, dedičnosť a podobne) a sú skúmané už niekoľko desaťročí. Ich nasadenie na niektoré problémy prináša výborné riešenia - napríklad v prípade infekčnosti vírusov, tvorby protilátok v imunitnom systéme a podobne - teda v mikroevolučnej škále. Ich rýchlosť a adekvátnosť v širšom - makroevolučnom merítku (teda nové orgány, tvorba novej, výhodnej, komplexnej informácie v biologických systémoch) je však vážne spochybňovaná.[14] Ako simulátory reálnych evolučných procesov sú kritizované z niekoľkých aspektov. V prvom rade prehľadávania priestoru riešení je veľmi neefektívne a pomalé, než aké vyžaduje vývoj v priemysle. Usmernenie laborantom býva často nevyhnutné viacráz. [15] Ďalej je vysoko pravdepodobné, že riešenia, ku ktorým nie je možné sa približovať postupne (tzv. rozoklané adaptívne povrchy prípadne rozhodovacie problémy so „skokovitou“ fitness funkciou), nebudú v „rozumnom“ čase nájdené vôbec.[16] [17]
Referencie
[upraviť | upraviť zdroj]- ↑ „Princípy smerovanej evolúcie“ http://www.chymiatrie.de/index.php/component/content/article/133-video-37 Archivované 2011-07-18 na Wayback Machine
- ↑ Arnold, FH; Wintrode, PL; Miyazaki, K; Gershenson, A (February 2001). "How enzymes adapt: lessons from directed evolution". Trends in Biochemical Sciences. 26 (2): 100–6. doi:10.1016/s0968-0004(00)01755-2. PMID 11166567 https://pubmed.ncbi.nlm.nih.gov/11166567/
- ↑ Behe, M.: Darwinova černá skříňka. Návrat domů, Praha. 2001.
- ↑ https://www.repository.cam.ac.uk/handle/1810/245207
- ↑ Gabriel L. Butterfield,1,2,3,* Marc J. Lajoie,1,2,* Heather H. Gustafson,4,5 Drew L. Sellers,4,5,6 Una Nattermann,1,2,7 Dan Ellis,1,2,3 Jacob B. Bale,1,2,3 Sharon Ke,4 Garreck H. Lenz,8 Angelica Yehdego,8 Rashmi Ravichandran,1,2 Suzie H. Pun,4,5 Neil P. King,1,2 and David Baker1,2 Evolution of a Designed Protein Assembly Encapsulating its Own RNA Genome Nature. 2017 Dec 21; 552(7685): 415–420. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5927965/
- ↑ Ann K Gauger, Douglas D Axe: The Evolutionary Accessibility of New Enzymes Functions: A Case Study from the Biotin Pathway. BIO-Complexity Vol 2011. http://bio-complexity.org/ojs/index.php/main/article/view/BIO-C.2011.1/BIO-C.2011.1
- ↑ John Reidhaar-Olson, Robert Sauer: Functionally Acceptable Solutions in Two Alpha-Helical Regions of Lambda Repressor. Proteins: Structure, Function, and Genetics 7, 1990, http://www.ncbi.nlm.nih.gov/pubmed/2199970
- ↑ Douglas Axe: The Limits of Complex Adaptation: An Analysis Based on a Simple Model of Structured Bacterial Populations. BIO-Complexity Vol 2010. http://bio-complexity.org/ojs/index.php/main/article/view/BIO-C.2010.4/BIO-C.2010.4
- ↑ E. Zuckerkandl, “The Appearance of New Structures in Proteins During Evolution,” p.21.
- ↑ Marcel Shutzenberger, “Algorithms and the Neo-Darwinian Theory of Evolution,” in P.S. Morehead and M.M. Kaplan, eds., Mathematical Challenges to the Darwinian Interpretation of Evolution, (Wistar Institute Symposium Monograph, 1967).
- ↑ Gauger, A., & Axe, D. (2011). The Evolutionary Accessibility of New Enzymes Functions: A Case Study from the Biotin Pathway. BIO-Complexity, 2011(0). http://bio-complexity.org/ojs/index.php/main/article/view/BIO-C.2011.1
- ↑ Romero PA, Arnold FH (2012) Random Field Model Reveals Structure of the Protein Recombinational Landscape. PLoS Comput Biol 8(10): e1002713. http://dx.doi.org/10.1371/journal.pcbi.1002713
- ↑ Douglas D. Axe. Estimating the Prevalence of Protein Sequences Adopting Functional Enzyme Folds. Journal of Molecular Biology, Volume 341, Issue 5, 27 August 2004, Pages 1295–1315. http://dx.doi.org/10.1016/j.jmb.2004.06.058
- ↑ Murray Eden: Inadequacies of Neo-Darwinian Evolution as a Scientific Theory. In: Mathematical Challenges to the Neo-Darwinian Interpretation of Evolution. 9-11. Wistar Institute Symposium Monograph. New York 1967
- ↑ Marcel Schützenberger: Algorithms and the Neo-Darwinian Theory of Evolution. In: Mathematical Challenges to Neo-Darwinian Theory of Evolution. 9-11. Wistar Institute Symposium Monograph. New York 1967
- ↑ Stuart Kauffman and Simon Levin, “Towards a General Theory of Adaptive Walks on Rugged Landscapes,” Journal of Theoretical Biology 128 (1987): 11-45. See also Stuart Kauffman, Origins of Order: Self-Organization and Selection in Evolution (Oxford: Oxford University Press, 1993), pp. 34-36;
- ↑ George Montañez, Winston Ewert, William Dembski, Robert Marks: A Vivisection of the ev Computer Organism: Identifying Sources of Active Information. BIO-Complexity Vol 2011. http://bio-complexity.org/ojs/index.php/main/article/view/BIO-C.2010.3
- Tento článok je čiastočný alebo úplný preklad článku Directed evolution na anglickej Wikipédii.
Pozri aj
[upraviť | upraviť zdroj]Literatúra
[upraviť | upraviť zdroj]- Behe, M.: Darwinova černá skříňka. Návrat domů, Praha. 2001.
- Arnold, FH; Wintrode, PL; Miyazaki, K; Gershenson, A (February 2001). "How enzymes adapt: lessons from directed evolution". Trends in Biochemical Sciences. 26 (2): 100–6. doi:10.1016/s0968-0004(00)01755-2. PMID 11166567
Externé odkazy
[upraviť | upraviť zdroj]- George Montañez, Winston Ewert, William Dembski, Robert Marks: A Vivisection of the ev Computer Organism: Identifying Sources of Active Information. BIO-Complexity Vol 2011. - [1]
- Romero PA, Arnold FH (2012) Random Field Model Reveals Structure of the Protein Recombinational Landscape. PLoS Comput Biol 8(10): e1002713. [2]
- „Princípy smerovanej evolúcie“ [3] Archivované 2011-07-18 na Wayback Machine
